カテゴリー:感染症とCOVID-19
カテゴリー:社会・時事問題
カテゴリー:ウイルスの話
新型コロナウイルスの感染症については、第2波、第3波、第4波...へ向けた対策が必要です。その中でも重要なのが、いかにして早く感染者を検査で見つけ出し、感染拡大を抑えるかということです。これまでの流行の過程で、COVID-19と原因ウイルスSARS-CoV-2に関するさまざまな科学的知見が得られてきましたが、その中には、あらたな監視システムの可能性に繋がる情報も得られています。
新しい監視システムの一つとして注目されているのが、下水を使ったウイルスのモニタリングです [1, 2, 3]。すでに海外から興味深い論文が次々と出されています。ここでは、下水のウイルス監視(サーベイランス)システムについての可能性を考えてみたいと思います。
下水の環境サーベイランスについては、すでにポリオウイルス [4] やノロウイルス [5] の例があり、また、メタゲノミクスによる抗生物質耐性遺伝子探索 [6] の先行研究があります。日本では、仙台市におけるノロウイルス監視システムの実例があります。
1. 下水中のウイルス検出
昨日、今日のテレビのニュースやワイドショーで、世界で行われている下水中のSARS-CoV-2の調査を紹介していました。新聞もこの話題を取り上げています [7]。しかし、日本ではまだ本格的に当該ウイルスの調査は始まっていないようで、テレビでは、分析方法の確立後に調査するような伝え方をしていました。今は、25の自治体が大学等に分析依頼に出した段階で、処理施設側は下水サンプルを凍結保存しているという状況らしいです。
私はこれらを視聴していて、もしそうであるなら、正直言って日本はずいぶん遅れているなと感じました。私は以前、民間会社の受託分析ビジネスで下水中のさまざまなウイルスの調査を行なっていましたので、それが比較的簡単に実施できることや、情報の重要性を理解しています。当時私が行なっていた操作手順を図1に示します。
下水そのままでは、ウイルスの濃度が薄くてPCRでさえも直接検出することは困難です。そこで、超遠心分離機を使って、あるいは市販の遠心フィルターユニットを通してウイルスを濃縮する必要があります。これらの方法では、大量のサンプルを扱うことは必ずしも容易でないので、後述するように、化学的処理で濃縮するのが有効かもしれません(オーストラリアの例)。
濃縮サンプルを少量のバッファー(緩衝液)に再懸濁し、ウイルスRNAを抽出します。そしてPCRにかけるために、抽出したRNAを酵素を使ってDNA(cDNA)に変えます。これを逆転写といいます。私が用いていたプロトコールでは、ここまでの作業で約2時間かかります。
PCRは機械が行うので楽ですが、ここでも1時間以上かかります。上記の逆転写したcDNAを鋳型として、リアルタイムPCR(RT-PCR)を行います。リアルタイムPCRとは、DNAの増幅過程がリアルタイムでモニタリングできるということから、このように呼ばれます。このPCRは半定量的ですが、きちんとウイルス量を測ることもできます(定量PCRといいます)。全体としてはそれぞれ、逆転写リアルタイムPCR(rRT-PCR)および逆転写リアルタイム定量PCR(rRT-PCR)と呼ばれます。
PCRでは、標的の2本鎖DNA領域を両側から挟み込むように複製していきますが、このためには、標的DNAの両端に特異的に結合する、プライマーと呼ばれる2本1組の短い合成DNA(オリゴヌクレオチドと言います)が必要です。SARS-CoV-2の検出用にもさまざまなプライマーセットが設計されていますが、その種類によって、増幅領域や増幅効率も異なりますので [8]、プライマーの選択が検出成功の一つの鍵になります。
そして現在SARS-CoV-2の検出使われている世界標準の検査法は、PCRの中でも最も精度の高いマルチプレックスプローブです。これは複数の領域を標的とするプライマーと蛍光プローブのセットを使うRT-PCRで、DNAにハイブリッドして消光状態にあったプローブが、DNAの増幅が起こるたびに分解されて蛍光を発するようになっています(ロシュの登録商標を付けて"TaqMan" PCRと呼ばれています)(→PCR検査をめぐる混乱)。
私が行なっていた頃はRNA抽出が比較的むずかしく、操作に熟練を要しました。今ではRNA抽出からPCRにかけるまで一つの市販キットで行うことができるようになっており、RT-PCR装置も非常に精度が高く、標準タイプで一度に96サンプルを分析できるようになっています。
ですから、分析方法の確立などはすぐにでもできるはずです。私は、COVID-19患者の肛門スワブ、糞便、尿にSARS-CoV-2が含まれるという論文 [9, 10, 11] が出た時に、すぐに下水のウイルス調査を行なうべきだと思いました。事実、海外では、2020年2月に入ると、すぐに下水のSARS-CoV-2調査が始まりました。
一方で、日本はだいぶ出遅れた感は否めません。日本水環境学会が今月プロジェクトを立ち上げ [12]、各自治体と大学が協力して下水の分析を始めている段階です。
2. 各国の下水ウイルスの調査
これまで、オランダ [1, 13]、フランス [14]、イタリア [15]、スペイン [16]、米国 [17, 18]、オーストラリア [19] などの研究グループが、下水中のSARS-CoV-2の分布について論文を発表しています。これらのいくつかについて、以下に簡単に紹介したいと思います。
2-1. オランダ
オランダの研究チームは、2020年3月29日、プレプリント論文集であるメドアーカイヴmedRxivに成果を発表しました [13]。調査対象としたのは、アムステルダムを含む7つの都市とスキポール国際空港の都市廃水処理場です。これらの処理場から2-3月に下水を採取し、遠心分離で大きな粒子を除去したのち、上澄みをCentricon® Plus-70 遠心フィルターユニットに通してウイルスを集めました。サンプル量は36–150 mLであり、比較的大きな量を使っています。
ウイルスの検出には、常法であるrRT-PCRを用いました。増幅する標的領域は、SARS-CoV-2のカプシドタンパクであるN1-3をコードする遺伝子、およびエンベロープタンパクEをコードする遺伝子です。ロシュ製のTaqMan PCRキットを使って、45サイクルのrRT-PCRを行なっています。
表1に示すように、オランダで最初の感染事例から3週間前の2月6日前後においては、ウイルスは検出されませんでした。しかし、3月5日には5カ所からN1遺伝子が検出されました。3月15/16日にはN1が6カ所から、そしてN3とEが、それぞれ5カ所および4カ所から検出されました。
表1. オランダの各都市の下水からのSARS-CoV-2の検出(文献[13]からの転載表に加筆)

これらの結果から、著者らは、感染の状況をあらかじめ知る上で下水のサーベイランス(監視)がよい手法になると示唆しています。ただ、選んだプライマーと標的領域によって検出効率が異なっていることには、注視すべきでしょう。
2-2. フランス
フランスの研究は、やはりメドアーカイヴに発表されています [14]。最初の報告は2020年4月12日でしたが、5月6日にアップデート版が出されています。この研究では、パリの3ヶ所の下水処理場から、3-4月に下水を採取し、その11 mLを超遠心分離機にかけてウイルスを集めています。SARS-CoV-2の検出には同様にrRT-PCRを使っていますが、こちらはウイルスのゲノム(RNA)を定量しているrRT-qPCRです。標的領域は、エンベロープのEタンパク質遺伝子です。
このフランスの研究チームの成果は、日本のテレビニュースでも紹介されました。その際に示されたものが図2です。

図2. テレビニュース(TBS 「News23」2020.05.29)で紹介されたフランスの研究結果.
図3には、実際の原著に示されている当該図を比べて示します。両者を比較するとわかりますが、テレビ(図2)で下水のウイルス量とされているのは、実際は有症状受診者や患者の数から予測されるウイルス排出量であり、PCRで検出されたウイルス量は、図3の三角印のグラフだということがわかります。
図3からわかるように、陽性患者が増加し始める1週間前から、下水中にウイルスが検出されていることがわかります。
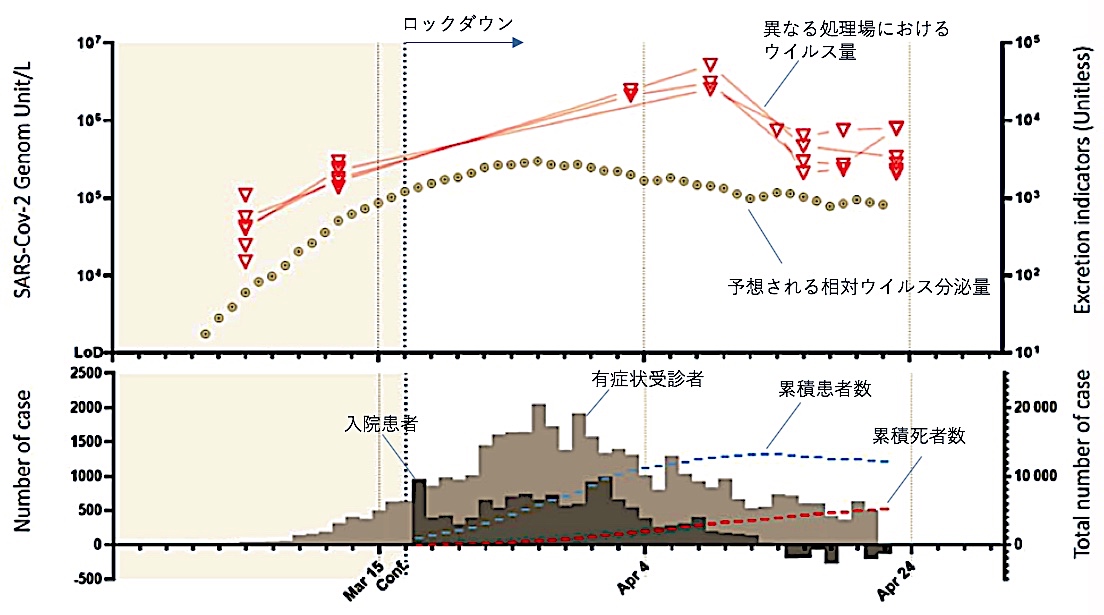
図3. フランスの研究グループによる下水中のSARS-CoV-2の調査ーCOVID-19患者数との関係(文献[14]からの転載図に加筆).
これらの結果から、下水監視システムは、個人のプライバシーなどの倫理的問題を生じることなく、経済的理由で個人の検査が困難な場合などに、患者発生に先駆けて感染状況を検知する方法として有用であり、とくに貧困国の流行におけるサーベイランスとしても活用できるとしています。
2-3. 米国
イェール大学の研究グループは、5月22日、研究成果をメドアーカイブに発表しました [17]。2020年3月19日から5月1日まで、コネチカット州ニューヘイブンの下水処理場から毎日下水汚泥サンプルを採取し、分析に用いました。サンプル量は2.5 mLとかなり小さく、直接市販のRNA抽出キットを用いてRNAを抽出しました。rRT-qPCRの標的は、N1とN2遺伝子であり、40サイクルで増幅しています。
図4に示すように、下水汚泥混液に含まれる当該ウイルスRNAの濃度は、時間差があったものの、感染者の流行曲線や医療機関の入院患者数と高い相関が認められました。そして、ウイルスRNAの検出は、新規陽性者の発生が起こる7日前、入院患者数が発生する3日前に、立ち上がりが見られました。

図4. イェール大学の研究チームによる下水汚泥混液中のウイルスRNA量の経時的変化: A、ウイルスRNA量(赤抜き丸)と入院患者数(黒抜き丸); B、ウイルスRNA量と入院患者数のリトレース・スムージング; C、ウイルスRNA量と新規感染者数(流行曲線); D、ウイルスRNA量と新規感染者数との関係; E、Dのスムージング(文献[17]から転載).
このようなウイルスの検出と感染者・入院患者の発生との間に時間差があるのは、症状が現れるまで検査しないためと考えられ、無症状感染者からのウイルス排出が、下水中に反映されているということになります。
これらの結果から、下水のウイルスモニタリングは、感染流行を予測し、予防策の強化や緩和をタイムリーに判断できる監視システムとして機能すると著者らは述べています。さらに、とりわけ検査体制が脆弱な発展途上国では、下水や汚泥の監視システムが役に立つだろうと指摘しています。これは、上述したフランスの研究チームが指摘していることと同じです。
米国ではまたMITの研究チームが、メドアーカイヴに研究成果を発表しています[18]。マサチューセッツ州の下水処理場から下水を採取し、スパイクタンパク質SとN1-N3を標的としてrRT-qPCRを行なっています。増幅されたN1-N3の遺伝子の違いでコピー数に若干ばらつきがありましたが、おおむね比例して検出されています。
増幅する領域の違いで検出効率が異なるというのは、前記したオランダの研究と似たようなところがあります。
この研究のおもしろいところは、サンプルの冷蔵保存の影響を調べているところです。4℃で1週間保存したのち、再度検出を試みた結果、1週間前とほぼ同様なコピー数が得られました。すなわち、4℃の冷蔵庫保存でも1週間はウイルスが壊れることなく保存できるということです。
2-4. オーストラリア
オーストラリアの研究チームも、メドアーカイヴに下水の調査結果を発表しています [19]。この研究では、クイーンズランドにある廃水処理場の下水を対象として調査しています。下水サンプルからのウイルスの濃縮には、方法Aでは陰電荷のメンブレンフィルターろ過を使い、方法BではCentricon® Plus-70 centrifugal filterを使っています。rRT-PCRの標的はNタンパク遺伝子であり、Cormanら [20] が開発したプライマーと日本の国立感染研究所(NIID)が開発したプライマー [21] を使っています。
結果として、下水中のウイルスは、NIIDのプライマーでは全く検出されず、Cormanのプライマーでは1サンプルだけ検出されたのみでした。その場合も、コピー数が1.9〜12 copies/100 mLと小さいものでした。
したがって、下水ウイルスのPCR検出は、軽症や無症状の感染者の感染拡大の初期のモニタリング手法として有望だと思われるものの、低い濃度では技法によって結果が異なり、低いウイルス濃度でさえも検出するような技法の改善が課題だとしています。
オーストラリアではもともと感染者数が少ないので、下水に流れ込んでいるウイルス量もきわめて小さく、検出限界ギリギリだった可能性があります。これは、サンプル量の工夫で解決できるのではないかと思われます。日本も欧米に比べると感染者数が著しく少ないので、同様な工夫が必要かもしれません。
しかしながら、特筆すべきは方法Aによるサンプルの前処理です。この方法では、サンプルを塩酸でpH 3.5–4に調整し、孔径0.45μmのメンブレンフィルター上に核酸粒子をトラップできるようにしている点です。これだと数百mLのサンプルを短時間で簡単に濃縮することができます。
3. 日本の対策の方向性
COVID-19感染に関する知見で最も注目すべきことは、不顕性感染(無症状者)からの二次感染が感染拡大の起点になっていることと、感染者の発症1日前が感染力が強いということです。そして、感染患者の治療に至る前の「検査と隔離」を徹底した国ほど(例:ヨーロッパにおけるドイツ、アジアにおける韓国、台湾、ヴェトナムなど)、感染拡大を抑え、そしてより低い死亡率と致死率を達成していることです。
これらの知見と現状を合わせると、流行の可能性をいかにいち早く察知し、かつスーパースプレッダーの可能性がある発症前感染者および無症候性感染者を探知して隔離するかということが重要なことがわかります。これが医療と経済活動への負荷を軽減することにも繋がります。つまり早い段階で隔離して治療すれば重症化→死亡という事例も少なくできますし、隔離が徹底できればロックダウンや接触機会削減も実施することなく、あるいはそれらを最低限にしながら、経済活動が維持できると言うことです。
この面で、上述したように、下水のウイルスのモニタリングは、感染流行を予測し、予防策の強化や緩和を適宜判断できる監視システムとして、効果的に機能する可能性があります。すなわち、区域ごとの下水を、住民全員の検体とみなして検査・監視していくというやり方であり、非常に安上がりかつ労力をかけないですむというメリットがあります。
従来の日本の従来の対策をみると、クラスター戦略の中で、具合が悪いという人たちの連絡を受けてからPCR検査で患者としての受け入れ順序を決め、かつ受診の目安というハードルのために、発症後1週間以上経過して重症化した人から(極論すれば、わざわざ重症化するまで待って)治療を開始するという、まったくの後手後手のやり方でした。つまり、前のブログ記事「世界が評価する?日本モデルの力?」でも指摘しましたが、病床数への圧迫を避けるために重症患者を優先的に選ぶことにより、かえって時間差で重症患者と死亡者を増やしてしまったのではないかと推察されます。
言い換えれば、日本においてはPCR検査を単に医療行為として限定使用したがために、かつ元々高齢化率が高いという要因もあって、さらに重症化と死亡する確率を高めてしまったのではないかということです。この可能性について検証が必要ですが、やり方を変える必要があることは論を待たないと思われます。その一つのオプションとして、医療資源のPCRという考え方に加えて、スクリーニングと監視システムの中のPCR(およびその他の検査)も考えるという方針への発想転換が、必要ではないかということです。
感染者を探し出すために、PCR検査を網羅的に行うというというのは理想的ですが、現状ではキャパシティーの問題があっていささか効率が悪いです。中国の武漢では全人口に対して検査を行うことが進められていますが(すでに900万人が検査済み)、こういう力技は今のところ日本では無理でしょう。この面で、下水のウイルスを監視するという方法は、感染予測法として簡便で効果的であり、日本にとっても採用しやすい方法だと考えられます。
ただ、中国のやり方は個人個人の検体を別々に検査するのではなく、一定数プールして一つの検体として供試する方法ですので、より短時間で済ませることができます。つまり、集団ごとに1検体として検査して、陽性が出た集団の中からさらに個々を精査するという賢いやり方です。考え方によっては、下水を監視するシステムと似たようなところがあります。
下水のウイルス監視システムのメリットは、地域ごとの下水処理場にそれを設ければ、ウイルスと感染状況に関する空間・地域分布を常時可視化できるのではないかと思われることです。つまり、現在行われている天気予報のような二次元空間分布としての使い方が可能であり、それによって感染予防のための、段階的警戒レベルの発出などの迅速な対応ができるのではないかと期待できます。また、地域や国別のより正確な流行・感染状況の可視化にも使えると考えられます。
現在日本での流行のパターンは、COVID-19患者の発症日ごとの数を積み上げたエピカーブによって推定されていますが、これは検査で確定された患者数に依存するという欠点があります。一方、下水監視の場合は、潜在的感染者をも含めた網羅的なプール分析になりますので、結果が検査数に依存するということがなく、実際に日本に存在するウイルス量に基づくより正確なエピカーブを描けるのではないかと思います。。
さらに下水の分析に適宜RNAメタゲノミクス(RNA転写物のゲノム解析)を使うことにより、ウイルスの変異パターンの程度と多様性を逐次知ることができるのではないかと期待できます。ゲノム解析で変異部分がわかれば、その部分を標的とするマルチプレックスプローブRT-PCRや標準RT-PCRの融解曲線解析によって、PCRの段階で変異を知ることもできます。
おわりに
下水のウイルス監視システムは、感染者が少ない場合はウイルス量が小さくなり、検出できなくなるなどの課題はありますが、その簡便性と経済性(低コスト)においてとても有望な方法だと考えられます。
また、下水からの感染のリスクもなお課題として残されていますが、有用な情報源としての活用のメリットの方が大きいと思われます [1]。
今までの日本の対策は、クラスター発生周辺の聞き込みや接触削減という、いかにもアナログ的・古典的なアプローチでありますが、アプリの活用とともに、検査を社会監視の攻めの武器として使う、先端的戦略に切り替える必要があるでしょう。すでにこの観点から、18道県知事は連名で、PCRの拡充とICTの積極的活用を提言しています [22]。
このような網羅的な感染監視システムを効果的に使うことができれば、経済的・生活被害の大きい大掛かりな接触削減は、最低限のレベルに抑えられる可能性があります。経済・社会活動の再開の指標としても、下水ウイルス量はきわめて有効であり、早急に主要都市を繋ぐ下水監視システムネットワークの構築が望まれるところです。
引用文献・記事
[1] Lodder, W. and de Roda Husman, A. M.: SARS-CoV-2 in wastewater: potential health risk, but also data source. Lancet Gastroentrol. Hepatol. 5, 533-534 (2020). https://www.thelancet.com/journals/langas/article/PIIS2468-1253(20)30087-X/fulltext
[2] Smriti Mallapaty: How sewage could reveal true scale of coronavirus outbreak. Nature 03 April 2020. https://www.nature.com/articles/d41586-020-00973-x
[3] 松岡由希子: 下水が新型コロナ早期警戒システムになる? Newsweek日本版. 2020.05.28. https://www.newsweekjapan.jp/stories/world/2020/05/post-93533.php
[4] Lodder, W. J. et al.: Feasibility of quantitative environmental surveillance in poliovirus eradication strategies. Appl. Environ. Microbiol. 78, 3800–3805 (2012). https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3346415/
[5] Hellmér,M. et al.: Detection of pathogenic Viruses in Sewage Provided Early Warnings of Hepatitis A Virus and Norovirus Outbreaks. Apple. Environ. Microbial. 80, 6771–6781. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4249052/
[6] Hendriksen, R. S. et al.: Global monitoring of antimicrobial resistance based on metagenomics analyses of urban sewage. Nat. Commun. 10, 1124 (2019). https://www.nature.com/articles/s41467-019-08853-3
[7] 香取啓介: 下水からウイルス、パリで検出 感染拡大の察知可能に? 朝日新聞DIGITAL 2020.04.24. https://digital.asahi.com/articles/ASN4R5RQBN4RUHBI005.html
[8] Vogel, C. B. F. et al.: Analytical sensitivity and efficiency comparisons of
SARS-COV-2 qRT-PCR primer-probe sets. medRxiv Posted April 26, 2020. https://www.medrxiv.org/content/10.1101/2020.03.30.20048108v3
[9] Zhang, W. et al: Molecular and serological investigation of 2019-nCoV infected patients: implication of multiple shedding routes. Emerg. Microbes Infect. 9, 386-389 (2020). https://www.tandfonline.com/doi/full/10.1080/22221751.2020.1729071
[10] Sun, J. et al.: Isolation of infectious SARS-CoV-2 from urine of a COVID-19 patient. Emerg. Microbes Infect. 9, 991–993 (2020). https://doi.org/10.1080/22221751.2020.1760144
[11] Chen, Y. et al.: The presence of SARS‐CoV‐2 RNA in the feces of COVID‐19 patients. J. Med. Virol. First published:03 April 2020. https://doi.org/10.1002/jmv.25825
[12] ⽇本⽔環境学会: ⽇本⽔環境学会COVID-19 タスクフォース設⽴のお知らせ. 2020.05.15. https://www.jswe.or.jp/pdf/COVID-19TF_JSWE.pdf
[13] Medea, G.: Presence of SARS-Coronavirus-2 in sewage. medRxiv Posted March 30, 2020. https://www.medrxiv.org/content/10.1101/2020.03.29.20045880v1
[14] Wurtzer, S. et al: Evaluation of lockdown impact on SARS-CoV-2 dynamics through viral genome quantification in Paris wastewaters. medRxiv Posted May 06, 2020. https://www.medrxiv.org/content/10.1101/2020.04.12.20062679v2
[15] Giuseppina La Rosa, et al.: FIRST DETECTION OF SARS-COV-2 IN UNTREATED WASTEWATERS IN ITALY. medRxiv Posted May 07, 2020. https://www.medrxiv.org/content/10.1101/2020.04.25.20079830v2
[16] Walter Randazzo et al.: Metropolitan wastewater analysis for COVID-19 Epidemiological Surveillance. medRxiv Posted April 29, 2020. https://www.medrxiv.org/content/10.1101/2020.04.23.20076679v2
[17] Peccia, J. et al. SARS-CoV-2 RNA concentrations in primary municipal sewage sludge as a leading indicator of COVID-19 outbreak dynamics. medRxiv posted May 22, 2020. https://www.medrxiv.org/content/10.1101/2020.05.19.20105999v1
[18] Wu, F. Q. et al.: SARS-CoV-2 titers in wastewater are higher than expected from clinically confirmed cases. medRxiv posted April 07, 2020. https://www.medrxiv.org/content/10.1101/2020.04.05.20051540v1
[19] Ahmed, W. et al.: First confirmed detection of SARS-CoV-2 in untreated wastewater in Australia: A proof of concept for the wastewater surveillance of COVID-19 in the community. Sci. Total Environ. Published online 18 April 2020, 138764. https://www.sciencedirect.com/science/article/pii/S0048969720322816
[20] Corman, V.M., et al. 2020. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 25 (3),2000045. https://www.eurosurveillance.org/content/10.2807/1560-7917.ES.2020.25.3.2000045;jsessionid=x05vR1q6kOPqKnUC5T7y_lLl.i-0b3d9850f4681504f-ecdclive
[21] Shirato, K., et al. 2020. Development of genetic diagnostic
methods for novel coronavirus 2019 (nCoV-2019) in Japan. Jpn. J. Infect. Dis. (2020) https://doi.org/10.7883/yoken.JJID.2020.061.
[22] 北海道知事ほか17県知事連名:感染拡大を防止しながら一日も早く経済・社会活動を正常化し、 日常を取り戻すための緊急提言. 2020年5月11日. https://s3-us-west-2.amazonaws.com/jnpc-prd-public-oregon/files/2020/05/dccea7ab-f94d-4c5d-ae2f-5e721e22d8a8.pdf
引用拙著ブログ記事
2020年5月26日 世界が評価する?日本モデルの力?
2020年3月24日 PCR検査をめぐる混乱
カテゴリー:感染症とCOVID-19
カテゴリー:社会・時事問題
カテゴリー:ウイルスの話
