カテゴリー:感染症とCOVID-19
はじめに
国立遺伝学研究所と新潟大学の共同研究チームは、新型コロナウイルス(SARS-CoV-2)のnsp14遺伝子が変異することが、第5派流行の減衰に繋がった可能性があるという研究成果を日本人類遺伝学会で発表しました。メディアは早速これを紹介しています [1]。
私はこの研究や学会発表の詳細を知りませんので、メディアの報道で類推するしかありませんが、どうやら、ゲノム複製のエラーをチェックし修復する酵素の遺伝子であるnsp14が変異し、ゲノム上の変異が蓄積して修復できず、ウイルスが死滅したということらしいです(図1)。

図1. メディアが報道した研究チームが考える酵素の変化がウイルスに与える影響([1]からの転載).
私はこの報道を見ていて違和感を感じました。もし報道どおりだとしたら、「変異して死滅するものが集団内で現存していた」ということ自体が生物やウイルスの進化の常識に反することだからです。致死的な変異が起こった場合、集団内に広がることはなく、速やかに排除され、適応したものだけが優勢になっていきます。自滅するような変異体はすぐに駆逐されてしまうので、私たちはそれを目にすることはできません。
SNS上では、いま話題の「エラーカタストロフ限界説」によるウイルス自壊と結びつけながら、早速この報道を取り上げて、流行減衰の理由付けに使っているようです。しかし、感染拡大抑制策とも関わることなので、根拠のない段階で断定することは危険です。
ここでは、焦点となっているnsp14遺伝子に着目して、その機能や変異の状況を文献を参照しながら紹介したいと思います。
1. 遺伝子の変異
まずは、ゲノムの変異と進化に関して、生物学の教科書に書いてあることを中心に復習してみます。
突然変異と進化を考える上で重要なのが分子進化の中立説です。この説では、生物やウイルスのゲノム変異はランダムに起こり、機能的にはよくも悪くもない中立であるとしています。つまり、あらゆる変異は方向性をもたないランダム変異であり、時間軸に対して確率的に起こるということです。これはゲノム複製の際のDNA(RNA)ポリメラーゼの複製ミスが一定の確率で起こるという内的要因に加えて、紫外線や放射線のような外的な変異原の影響が常にあるからです。
遺伝子が転写されタンパク質が作られる場合、元の塩基配列が三個単位(三つ組みコドン [codon])で読まれ、それが一つのアミノ酸に対応して翻訳されます。TTT GCT AGC という配列であった場合、フェニルアラニン(F)、アラニン(A)、セリン(S)という順に翻訳されます(図2)。ここで、コドンの3番目のCがTに置換された場合は(AGC→AGT)、その翻訳はやはりセリンで変化がありません。一方、AGC→ACCに置換された場合、翻訳はトレオニンに変わります。

図2. コドン(塩基配列の翻訳単位)における同義置換と非同義置換.
上述したように、遺伝子の塩基配列レベルで変異が起こった場合、アミノ酸の翻訳に変化を与えない場合を同義置換、翻訳に変化を与える場合を非同義置換といいます。一般的に、コドンの3番目の塩基が置き換わると同義置換となり、1、2番目の塩基が変わると非同義置換になります。
アミノ酸の置換が1個でも起こった場合、その重合体であるタンパク質の立体構造や機能に大きな影響を及ぼすことがあります。しかし、非同義置換の場合でも、極性や大きさなどにおいて性質が類似するアミノ酸への置換であれば、タンパク質の機能にほとんど変化を与えないこともあります。
したがって、ゲノムレベルでの変異(遺伝子変異)と一口にいっても、アミノ酸が変化せずまったく機能に影響しない場合、アミノ酸が置換しながら機能は(よくも悪くも)保たれる場合、機能が損なわれる場合の三つに大別されることになります(図3)。

図3. 同義置換と非同義置換がタンパク質の機能に与える影響.
このように、ゲノムレベルでの変異が機能に与える影響は、よくも悪くも、一様ではないことがわかります。そして、生物もウイルスも現存する時点では進化上最適化されているので、表現型に影響がある変異は一般に有害な場合が多く、ダーウィンの進化論にあるように自然淘汰されて行きます。これらの有害な変異を起こした個体は、その時点で消失しますので、私たちは目にすることができません。
要約すると、中立的な分子進化とダーウィンの自然淘汰によって、常に競争に勝ち、環境に適応したものが生き残り続けるということになります。ただし、後述するように、ウイルスは宿主との相互作用があり、宿主の抗ウイルス活性がウイルスの変異と進化に大きな影響を及ぼしていることを考慮する必要があります。つまり、分子進化の中立説には従わない偏った変異が起こっている可能があります。
SARS-CoV-2は宿主細胞内に侵入した後、脱殻というプロセスで自身のゲノム(1本鎖RNA)を放出します。このRNAが直接メッセンジャーRNA(mRNA)の役目をし、宿主細胞のリボゾーム上で翻訳され、ウイルスのタンパクが生産されます。一方、ウイルスのRNA複製は宿主細胞が行なうのではなく、作られた特定のウイルスタンパク質(RNA依存性RNAポリメラーゼ、RdRp)によって媒介されます。
RdRpは、nsp7, nsp8, nsp12、および nsp14 の4種類の非構造タンパク(non-structural proteins、nsp)で構成され、いずれもOrf1abの成熟タンパク質への分割で生成されます(総説参照 [2])。これらの中で、nsp12 が複製酵素(RNAポリメラーゼ)の本体であり、nsp14がエラー校正エキソヌクレアーゼ(ExoNase)活性およびグアニン-N7-メチルトランスフェラーゼ (N7-MTase)活性を有するタンパク質です [3]。
このようにSARS-CoV-2のRdRpにはエラー校正機能があるため、一見、頻繁に変異を起こすことはないように思われます。しかし、実際の変異頻度は校正機能がない他のRNAウイルスと大して変わりません。これは感染に抵抗するための宿主細胞のデアミナーゼによるRNA編集機能があるためと説明されています。すなわち、ウイルスRNAのアデノシン(A)がイノシンへ変換されたり、シトシン(C)からウラシル(U)へ変換されることが確認されており、このRNA編集プロセスによってウイルスと宿主の両方の運命が決定される可能性が指摘されています。
シチジンデアミナーゼであるAPOBEC(アポリポプロテインRNA編集酵素)ファミリーのメンバーは、RNA (DNA) ウイルスゲノムのC→U変換を媒介し、哺乳類細胞ではこの致死的編集による抗ウイルス活性があると考えられています [4, 5]。実際に、データベースにあるSARS-CoV-2のゲノム配列においては、プラス鎖RNAにおいてC→U方向の変異が非常に多く見られますが(後述)、このAPOBECファミリータンパクの作用の痕だと思われます。
もし、APOBECによってnsp14の機能に損なうような変異が起これば、ウイルスは増殖できなく可能性があります。すでに、SARS-CoV-2のExoNaseノックアウト変異体は、ゲノム複製できないことが報告されています [3]。ちなみに、以前のブログ記事で、APOBECによる抵抗ウイルス機能は、ファクターXの候補の一つとして挙げています(→日本の新型コロナの死亡率は低い?、COVID-19を巡るアジアと欧米を分ける謎の要因と日本の対策の評価)。
このように、SARS-CoV-2の進化(変異)は、自身のRdRpの複製エラー、nsp4のエラー校正と機能変化・消失、そして宿主との相互作用を組み合わせた結果ということになり、内的要因は複雑です。
SARS-CoV-2はベータコロナウイルスですが、nsp14の役割については、アルファコロナウイルスの先行研究があります。すなわち、そのExoNase活性を介して、宿主とウイルスの相互作用を調節し、ゲノム複製時に生成される二本鎖RNAを分解して免疫反応を抑制することで、ウイルスの存在率を高めることが示されています [6] 。
最近、宿主の翻訳の阻害因子としてのnsp14の機能が明らかにされています。病原性ウイルスが感染を成立させるためには、I型インターフェロン(IFN-I)の抗ウイルス反応を克服する必要がありますが、nsp14はIFN-I反応を阻害するというのです。米国イェール大学の研究グループは、SARS-CoV-2が宿主のタンパク質合成を停止させることができ、nsp14がこの活性を発揮することを報告しました [7]。
この研究によると、SARS-CoV-2 nsp14のExoNaseおよびN7-MTaseの活性部位に変異があると、その翻訳阻害活性が消失しました。さらに、nsp14-nsp10複合体が形成されると、nsp14による翻訳阻害作用が増強されました。その結果、IFN-Iに依存したインターフェロン刺激遺伝子(ISGs)の誘導が阻害されることがわかりました。以上のことから、SARS-CoV-2が翻訳阻害剤を介して宿主の自然免疫応答をシャットダウンしていると考えられます。
3. nsp14の変異はゲノムワイドの変異を増発する
トルコの研究グループ Eskierら [8] は、SARS-CoV-2ゲノムの変異率を高める原因として、nsp14が関係していること、そして多くの変異がC→U(論文ではDNAレベルでC→Tと表記)であることをPeerJ誌に報告しています。この報告をここで紹介したいと思います。
Eskierらは、SARS-CoV-2の変異頻度の経時的な傾向を明らかにするため、データベースにあるウイルスの全分離株について、全ゲノム、S遺伝子、M遺伝子、E遺伝子領域の1日あたりの平均変異密度を算出しました。この算出においては、異常値を95パーセンタイル値と5パーセンタイル値で除去し、シーケンスエラーによる潜在的な影響を最小化しています。M/E遺伝子領域の変異の有無は、ウイルス変異の頻度を探るよい指標とされており、著者らはこの論文でMoE statusと呼称しています。
変異密度の算出の結果、ゲノムレベル(図1A)とS遺伝子(図1B)の両方において、平均変異密度と時間の間に非常に強い正の相関が見られました。それに比べて、M/Eは弱い正の相関があり(図1C)、ゲノムやS遺伝子に比べて、初期と後期で平均密度の広がりが大きいことが分かりました。これは、既往の報告のように、M遺伝子とE遺伝子の選択圧が低下していることと一致するとしています。
ここでゲノム全体の変異密度とS遺伝子のそれを比べてみると、後者の方が傾きが大きいことが分かります。つまり、ゲノム全体に比べて、スパイクタンパク遺伝子の変化の許容度が高く、見かけ上、速く進化しているということです。
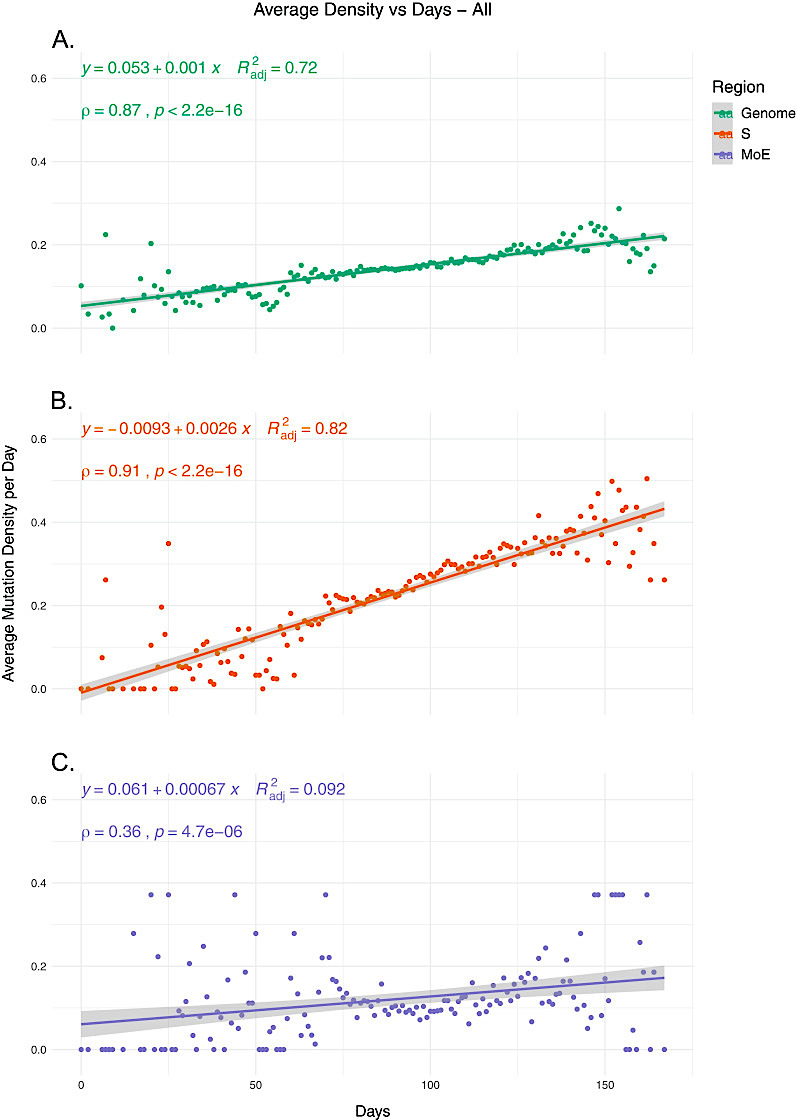
図4. SARS-CoV-2の進化速度. (A) ゲノム全体の突然変異密度と時間の関係、(B) S遺伝子の突然変異密度と時間との関係、(C) M遺伝子およびE遺伝子の変異密度と時間の関係 (文献 [7] から転載). Y軸の値は、対象領域のキロベースで正規化した、対応する日のSNVの平均数を表す. ゲノムのSNV数は、潜在的なシーケンスやアセンブリのアーチファクトの影響を最小限にするために、25パーセンタイルと75パーセンタイルの値でキャップして正規化. 相関スコアは、スピアマン順位相関Spearman rank correlationを用いて算出.
非同義的な変異としては、スパイクタンパク質のD614G置換を引き起こす23403 A→G(22,271株)が最も多く、次いで、RdRpタンパク質のP323L置換を引き起こすOrf1ab遺伝子nsp12領域の14408 C→T変異(22,226株)、Orf8タンパク質のL84S置換を引き起こす28144 C→T変異(3,081株)が見られました。
一方、最も多い同義的な変異は8782 C→T変異(3,047株)であり、これはOrf1ab遺伝子のnsp4コード領域に見られるものです。S遺伝子においては、最も頻度の高い同義的変異は23731 C→T変異(622株)でした。また、前述のD614G変異に次いで多い非同義的変異は、P1263Lの置換をもたらす25350 C→T(215株)でした。M/Eでは、最も多い同義的変異は26735 C→T(341株)、非同義的変異は27046 C→T(530株)で、いずれもM遺伝子に存在し、後者はT175Mのアミノ酸置換を引き起こします。D614G変異以外の変異はすべてC→T置換であり、この傾向はこれまでにも報告されています [9]。
図4に見られるように、機能的に影響の大きい遺伝子であるS遺伝子やOrf1abにおける時間の経過に伴う変異頻度は、M/Eなどの構造遺伝子に比べて顕著であることがわかります。そこで、著者らはSARS-CoV-2ゲノム(RNA)の複製に関与するこれらのタンパク質の変異が、変異増加と関連しているかどうかを検討しました。
Orf1abポリプロテインから切断されて生成する4つのタンパク質(nsp7、nsp8、nsp12 [別名RdRp]、nsp14)について、最も頻繁に観察される5つの変異を特定し、続いて、各変異とMoE statusとの関連をカイ二乗検定で分析しました。その結果、20個の変異のうち、合計12個の変異がMoE statusと有意に関連していることがわかりました。nsp14には4つの有意な変異が見られ、nsp7には2つ、nsp8には1つの変異が見られました。
nsp14の3つの変異(18060 C→T、18736 T→C、18877 C→T)は、すべてMoE statusと有意な関連を持ちますが、同様にゲノムの変異密度とも関係をもつことがわかりました(図5)。しかし、変異の部位でこれらとの関連性は異なりました。
たとえば、18060 C→T(L7L)は、MoE statusに対しては、野生型分離株に比べて同義的置換の変異密度の増加が遅く(図5A)、一方で、非同義的置換の変異密度増加の速さに大きな影響を与えていることがわかりました(図5B)。これに比べて、18877 C→T (L270L) は、どちらの置換においても変異密度が増加していることがわかりました(図5C, D)。
一方で、nsp7とnsp12の変異は、変異密度の増加率の変化にあまり影響を与えていないと著者らは述べています。

図5. 野生株(青色)と比較したnsp14変異を持つ分離株(赤色)における同義的変異(左)と非同義的変異(右)の分布(文献 [7] から転載). (A, B)同義的な18060 C→T 変異を持つ分離株 (n = 1,585); (C, D)同義的な18877 C→T 変異を持つ分離株 (n = 893); (E, F) 非同義の18736 T→C 変異を持つ分離株 (n = 236). すべてのグラフの野生型分離株は、9つの位置(11916, 12073, 13536, 13730, 13862, 14408, 18060, 18736, 18877)の参照ヌクレオチドを持っている(n = 5,910). 相関スコアは、スピアマン順位相関を用いて算出.
5. 感染流行とnsp14変異
nsp14の5つの最も一般的な変異のうち、18060 C→T、18736 T→C、18877 C→Tの3つの変異は、ゲノム全体の変異量と、ウイルス進化の代替指標であるMoE statusの両方の増加に関連していることがわかりました。しかし、18736 T→C のみが非同義変異(F233L)であるので、nsp14変異がウイルスの複製プロセスに及ぼす影響を理解するためにはより詳細な機能的研究が必要になると著者らは述べています。
nsp14の3つの変異は地域的な流行と関係があるようです。18060 C→T 変異は、2020年1月中旬に米国ワシントン州で検出された最初の症例に存在し、これ以降ほぼ米国に限定されています。一方、18877 C→T 変異は、サウジアラビアで1月末頃に発生した可能性が高く、検出された症例は少ないが、サウジアラビア(54.1%)とトルコ(37.4%)で最も多く見られたとしています。
18736 T→C 変異は、2020年3月初めに米国で初めて検出され、18060 C→T変異と同様に、ほぼ完全に米国に限定されています。他の2つとは異なり、この非同義的変異は2020年7月1日以降は検出されていないようです。異なるnsp14変異の優位性や消失は、ゲノム上の特定の変異とはあまり関係がないようですが、これらのnsp14変異がSARS-CoV-2の感染力に影響を与える可能性を否定することはできないと著者らは述べています。
SARS-CoV-2のコドン使用量をウイルスゲノムの時間進化の観点から分析した最近の研究では、nsp14が、S遺伝子およびN遺伝子とともに、最も高いCAI(Codon Adaptation Index)値を示す遺伝子の1つであることが明らかになっています [10]。CAI値は、最適なコドン使用の指標であり、どれだけ宿主に適応しているかを示すものです。
nsp14においてCAI値が高いことから、SARS-CoV-2にとって最適な突然変異率に達するために、このような突然変異が優先的に蓄積されてきたと推測できると著者らは述べています。先行研究の結果では、SARS-CoV-2ゲノムの変異密度はパンデミックステージと密接に関係しており、人口動態がウイルスゲノムの平均変異蓄積に直接影響するとされています。
英国や米国で3月に観測されたような急速な流行拡大では増殖が促進されます。この場合、複製忠実度をトレードオフにして、より高い複製率とより広い変異多様性を得ることができます。しかし、著者らも指摘していますが、複製装置に変異が起こり、全体の変異率が高くなりすぎると、有害なものとして排除される可能性が高くなます。
一方で、ごく一部の突然変異は、免疫逃避や抗ウイルス剤への耐性を付与するなど、有利に働く可能性もあります。著者らは今のところ、そのようなウイルスにとって有利な変異を検出することはできなかったと言っていますが、変異率が高くなると、そのような変異が現れる可能性が当然高くなります。
おわりに
SARS-CoV-2のnsp14タンパクは、ExoNaseおよびN7-MTaseの活性を有し、ウイルスのゲノム複製と宿主の抗ウイルス活性の阻害という重要な働きをしていることがわかってきました。そして、nsp14に重大な変異が起こるとウイルスに致死的に働くということも示唆されています。
COVID-19のパンデミック以来、各国、各地域で流行をもたらしているSARS-CoV-2にC→U変異が多く見られるということは、多数の宿主内でウイルス複製が起こる過程で、宿主APOBECファミリーによるRNA編集が頻繁に起こっていることを示唆しています。この活性がnsp14にも働き、nsp14変異株が各国・地域の流行に関わっていることは、Eskierら [8] の報告にあるとおりです。
nsp14を含めた複製装置に変異が起これば、その機能を損なう方向であれば、ウイルスは即座に消える運命にあるでしょう。変異し過ぎると排除されてしまうということになります。とはいえ、このような変異ウイルスは直ぐに消失しまいますから、株として多数検出されたとすれば、集団内で広がっていたことを示すものであり、広がる時点では致死的ではなかった、感染伝播する力があったということになります。事実、国立遺伝研と新潟大の結果は、実際の"自滅していない"ウイルス分離株で得られています。
その意味で、「nsp14が変異し、ゲノム上の変異が蓄積して修復できず、ウイルスが死滅した」という仮説は、ウイルスの進化と生態から考えれば矛盾します。多数の異なる感染者の中で、コピーミスによる致死的変異が同時に、あるいは次々と短時間で起こるとは考えにくいからです。むしろ、第5波流行ではnsp14変異デルタ株が蔓延し、それに対して宿主のRNA編集機能(抗ウイルス活性)が急速に効果的に働いたとした方が考えやすいです。つまり、ウイルスの自滅ではなく宿主側からの攻撃による壊滅です。
冒頭の仮説を第5波流行減衰の原因とするためには、少なくとも流行初期から収束するまでのウイルス分離株のゲノムを比較し、非同義的変異の蓄積度を明らかにし、その変異がnsp14タンパクの機能に阻害的になっていたことを、生理学的に時系列的に証明する必要があります。
引用文献・記事
[1] 共同通信:ゲノム変異、修復困難で死滅? コロナ第5波収束の一因か. Yahooニュース.2021.10.30. https://news.yahoo.co.jp/articles/ffc3131185430e24ea85bf09f4f8181e7ab3fa24
[2] 鈴木善幸: 重症急性呼吸器症候群2 (SARS-CoV-2) の起源と進化. 生物の科学 遺伝 75, 34–42 (2020). https://www.nsc.nagoya-cu.ac.jp/~yossuzuk/MyPapers.dir/34.pdf
[3] Natacha, S. et al.: The enzymatic activity of the nsp14 exoribonuclease Is critical for replication of MERS-CoV and SARS-CoV-2. J. Virol. 94, e01246-20 (2020). .https://doi.org/10.1128/JVI.01246-20
[4] 村松正道ら: B型肝炎ウイルスとAPOBECファミリー. 生化学 88, 557–562 (2016). https://seikagaku.jbsoc.or.jp/10.14952/SEIKAGAKU.2016.880557/data/index.html
[5] Ratcliff, J. & Simmonds, P.: Potential APOBEC-mediated RNA editing of the genomes of SARS-CoV-2 and other coronaviruses and its impact on their longer term evolution. Virology 556, 62-72 (2021). https://doi.org/10.1016/j.virol.2020.12.018
[6] Becares M. et al.: Mutagenesis of coronavirus nsp14 reveals its potential role in modulation of the innate immune response. J. Virol. 90, 5399–5414 (2016). https://doi.org/10.1128/JVI.03259-15
[7] Chun-Chieh Hsu, J. et al.: Translational shutdown and evasion of the innate immune response by SARS-CoV-2 NSP14 protein. Proc. Natl. Acad. Sci. USA 118, e2101161118 (2021). https://doi.org/10.1073/pnas.2101161118
[8] Eskier, D. et al. 2020. Mutations of SARS-CoV-2 nsp14 exhibit strong association with increased genome-wide mutation load. PeerJ 8, e10181 (2020). https://doi.org/10.7717/peerj.10181
[9] Simmonds, P.: Rampant C→U hypermutation in the genomes of SARS-CoV-2 and other coronaviruses: causes and consequences for their short-and long-term evolutionary trajectories. mSphere. 5, e00408-20 (2020). https://doi.org/10.1128/mSphere.00408-20
[10] Dilucca M, et al.: Temporal evolution and adaptation of SARS-CoV-2 codon usage. bioRxiv. Posted Jan. 22, 2021.
引用したブログ記事
2020年5月18日 COVID-19を巡るアジアと欧米を分ける謎の要因と日本の対策の評価
2020年5月13日 日本の新型コロナの死亡率は低い?
カテゴリー:感染症とCOVID-19